Sisukord:
- 1. samm: eeltingimused
- 2. etapp: eeltingimused (ei saa)
- 3. samm: eeltingimused (ei saa)
- 4. samm: eeltingimused (ei saa)
- Samm: puhastage Matlab, et valmistuda koodi käitamiseks
- Samm: valige 10 tavalist silmapilti ja 10 diabeetilise retinopaatia sümptomitega pilti
- Samm: valige 10 tavalist silmapilti ja 10 diabeetilise retinopaatia sümptomitega pilti (ei saa)
- Samm: looge 2 muutujat (tavaline ja diagnoositud) ja seadke need võrdseks 0 -ga
- 9. toiming: looge tsükkel tavaliste piltide automaatseks üleslaadimiseks
- 10. toiming: looge tsükkel tavaliste piltide automaatseks üleslaadimiseks (ei saa)
- 11. samm: kärpige pildi piire
- 12. samm: looge halli skaala pilt
- Samm: looge kontrastne pilt
- 14. samm: täiustage kontrastsust
- 15. samm: looge keskmine filter
- 16. samm: kombineerige keskmistusfiltri kontrastset pilti
- 17. samm: tehke pikslite lahutamisega uus keskmine mask
- 18. samm: looge binaarselt filtreeritud pilt
- Samm: eemaldage filtreeritud piltidelt leitud väiksemad plekid
- Samm: looge ketta struktureerimise element
- Samm 21: Tehke morfoloogilisi sulgemisoperatsioone
- Samm: leidke vähemalt 8 ühendatavad objektid
- Samm: leidke ühendatud pikslite maksimaalne arv
- 24. samm: määrake maksimaalsed piksliväärtused väärtusele 0 ja otsige piksleid, mille ühenduvus on> = 26
- 25. samm: eemaldage pildilt veresooned
- 26. samm: joonise kuvamine
- Samm: eemaldage anumad ja loendage vereplekid
- 28. samm: diagnoosige võrkkesta pilt tuvastatud verehüüvete arvu alusel
- Samm: kui on rohkem kui 5 plekki…
- Samm: korrake filtreerimisprotsessi normaalsete piltide jaoks, mille pildinumbri väärtused on 2 ja 3
- Samm: korrake diagnoositud piltide jaoks kogu protsessi
- 32. etapp: statistiline analüüs
- Samm 33. Usaldusintervalli leidmine
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 08:49.
- Viimati modifitseeritud 2025-01-23 14:40.
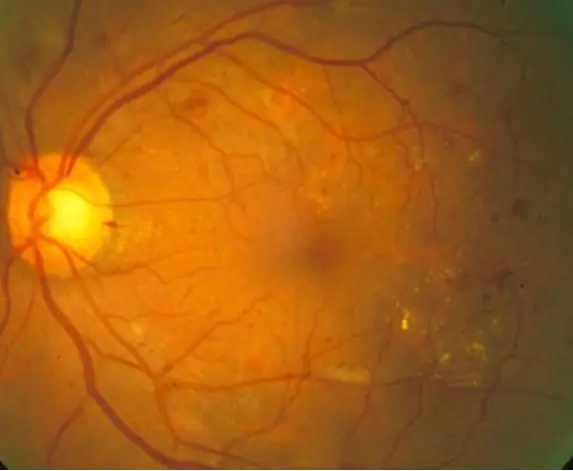

(Vaadake ülaltoodud koodi ülevaadet)
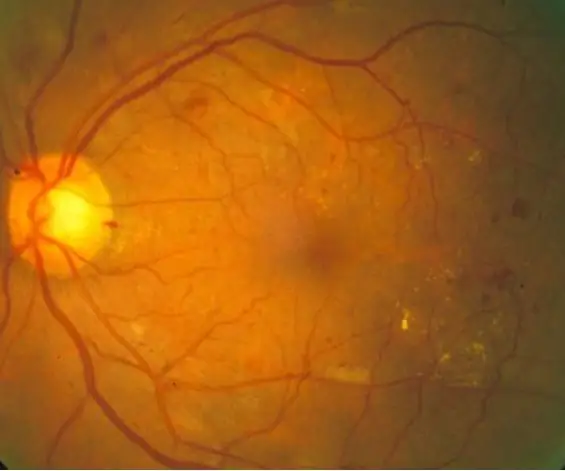
Diabeetiline retinopaatia on diabeediga seotud silmahaigus, mida põhjustab kõrge veresuhkru tase. Kõrge veresuhkru tase põhjustab võrkkesta veresoonte turset, mis põhjustab veresoonte laienemist ja isegi veresoonte lekkeid, mis põhjustavad võrkkesta piltidel tumedaid laike. Selle koodiga püüame kasutada veresoonte lekkekohtade väljanägemist diabeetilise retinopaatia taustal, kuigi reaalses maailmas oleks vaja täiendavaid diagnoosimismeetodeid. Selle koodi eesmärk on automatiseerida võrkkesta piltide töötlemist ja diagnoosimist, et tuvastada diabeetilise retinopaatia tunnused võrkkesta piltide tumedate laikude kaudu.
10 normaalset võrkkesta pilti ja 10 diagnoositud võrkkesta pilti töödeldi koodi abil, mis esmalt loeb ja filtreerib pilte ning seejärel kvantifitseerib tumedad laigud, et teha kindlaks, kas diabeetilise retinopaatia sümptomid esinevad antud läve alusel. Seejärel prinditakse tulemused vaataja tõlgendamiseks käsuaknasse.
1. samm: eeltingimused
1. Veenduge, et arvutisse oleks alla laaditud programm MATLAB.
2. Laadige alla lingilt leitud txt -fail. (MATLAB -koodiga samasse kataloogi salvestamiseks vajutage klahvikombinatsiooni ctrl+s)
2. etapp: eeltingimused (ei saa)
4. Avage MATLAB ja tippige käsuaknasse „uiimport”.
5. Valige fail officialdiagnoses.txt ja importige see lahtrimaatriksina MATLAB -i.
6. Veenduge, et näeksite tööruumis muutujana „ametlikud diagnoosid”.
3. samm: eeltingimused (ei saa)

7. Laadige alla funktsioon ModWald.m, mille saate ülaltoodud koodist või saate selle lõuendilt alla laadida.
(Koodi esitasid professor King ja professor Choi)
4. samm: eeltingimused (ei saa)

8. Laadige The STARE projekti andmete jaotisest alla 400 töötlemata pilti.
Samm: puhastage Matlab, et valmistuda koodi käitamiseks

Lisage koodile:
1. sulge kõik (sulgeb kõik varem avatud pildid)
2. clearvars - välja arvatud ametlikud diagnoosid (kustutab kõik muutujad, välja arvatud varem imporditud ametliku diagnoosi txt -fail)
3. cclc (tühjendab käsuakna)
Samm: valige 10 tavalist silmapilti ja 10 diabeetilise retinopaatia sümptomitega pilti
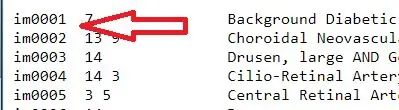
1. Võtke diagnoosi tekstifail ja eraldage piltide nimed. Need nimed on tekstifaili esimeses veerus, nii et nende ekstraheerimiseks sisestage „officialdiagnoses (:, 1)”. Pildinimede maatriks määrati muutujale „all_image_numbers”
2. Teisendage muutuja all_image_numbers lahtrimassiivist maatriksmassiiviks, kasutades funktsiooni cell2mat
Samm: valige 10 tavalist silmapilti ja 10 diabeetilise retinopaatia sümptomitega pilti (ei saa)
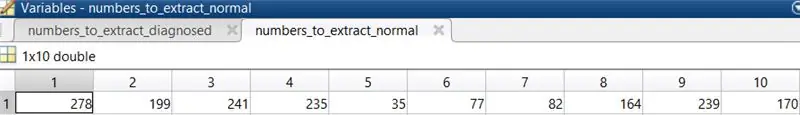
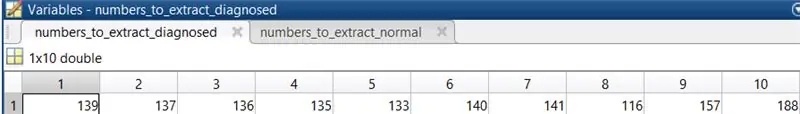
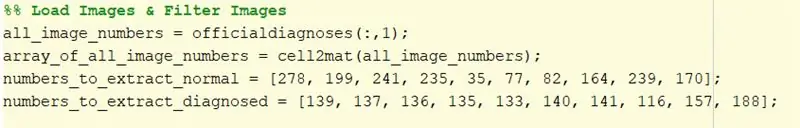
3. Valige koodi käitamiseks 10 tavalist silmapilti. Sel juhul valiti 278, 199, 241, 235, 35, 77, 82, 164, 239, 170.
Asetage need numbrid maatriksisse ja määrake need muutujale, millele helistatakse piltide laadimisel.
4. Korrake sammu 3 võrkkesta piltide puhul, millel on diagnoositud diabeetiline retinopaatia. Sel juhul valiti pildid 139, 137, 136, 135, 133, 140, 141, 116, 157, 188.
Samm: looge 2 muutujat (tavaline ja diagnoositud) ja seadke need võrdseks 0 -ga

Loo need muutujad enne silmust for, et lähtestada silmuste numbrid.
9. toiming: looge tsükkel tavaliste piltide automaatseks üleslaadimiseks

1. Loo for loop
2. Määrake loendamismuutuja (sel juhul i) väärtuste 1-10 maatriksile. Seda loendamismuutujat kasutatakse iga pildi eraldi helistamiseks
3. Võtke piltide maatriksis i -element, et eraldada ja teisendada pildinimi stringist numbriks, kasutades funktsiooni num2str.
Leidke numbrifunktsiooni abil pildinimes sisalduvate numbrite arv. Määrake see väärtus muutujale, digits_normal. See number peaks olema 1 ühekohaliste numbrite puhul, 2 kahekohaliste numbrite puhul ja 3 kolmekohaliste numbrite puhul. Seda teavet kasutatakse piltide automaatseks helistamiseks.
10. toiming: looge tsükkel tavaliste piltide automaatseks üleslaadimiseks (ei saa)

3. Looge if -lause, mis sisaldab kõiki kolme eelmiste sammude võimalust. Kui pildi nimi on 1 -kohaline, nimetatakse pilti kui „im000”, kui see koosneb kahest numbrist, siis pilti kui „im00” ja kui see on 3, nimetatakse pilti „im0”.
4. Iga if -lause all määrake muutujale imread “im” vastava, if -lause all oleva vastava arvu nullidega (nagu eespool kirjeldatud), millele järgneb i.
11. samm: kärpige pildi piire
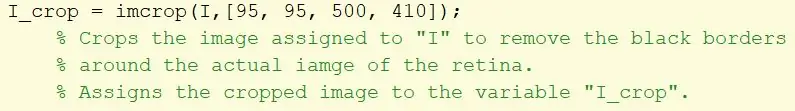
Võtke algne pilt ja rakendage mustade ääriste kõrvaldamiseks imcrop -filter ning määrake muutujale I_crop. Kärbitud ristkülik määratakse maatriksi abil [95, 95, 500, 410].
12. samm: looge halli skaala pilt

Võtke kärbitud pilt ja rakendage pildi halltoonide muutmiseks rbg2hall filter. Määrake see pilt muutujale I2.
Samm: looge kontrastne pilt
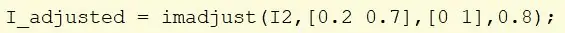
Tehke pilt I2 ja kasutage intensiivsuse väärtuste skaleerimiseks kohandust.
Võtke väärtused, mis jäävad vahemikku [0,2, 0,7] ja skaleerige need väärtuseks [0, 1]. Pildi heledamaks muutmiseks on gamma seadistatud väärtusele 0,8. Määrake uus pilt pildile I_adjusted.
14. samm: täiustage kontrastsust
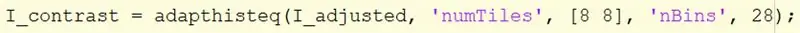
Võtke I_adjusted pilt ja kasutage kontrasti suurendamiseks adapthisteq funktsiooni.
Adapthisteqi süntaks nõuab pildi nime, I_adjusted, „numTiles”, numTiles suurust, „nBins” ja prügikastide arvu. Numbrite suurus on seatud [8 8], jagades pildi 8x8 plaatideks ja prügikastide arvuks 28. Määrake pilt I_constrast.
15. samm: looge keskmine filter

Looge muutuja nimega "meanfilt", kasutades funktsiooni fspecial. Sisestage keskmine funktsioon, et luua keskmistamise filter ja sisestage [90 90] lükandakna suurusele.
16. samm: kombineerige keskmistusfiltri kontrastset pilti
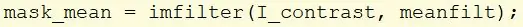
Looge uus muutuja nimega mask_mean ja kasutage funktsiooni imfilter, et teha pilt I_contrast ja rakendada varem loodud keskmine filter.
17. samm: tehke pikslite lahutamisega uus keskmine mask

Looge muutuja nimega mask_mean2 ja kasutage funktsiooni imsubtract, et lahutada I_contrast iga piksli väärtus maski_mean vastavast pikslist.
18. samm: looge binaarselt filtreeritud pilt

Muutke halltoonides kujutised mustvalgeks, kasutades imbinarize. Sisendmask_mean2, „adaptiivne”, „Esiplaani polaarsus”, „tume”, „Tundlikkus”, 0,6. Määrake see uus pilt mask_binarize'ile.
Samm: eemaldage filtreeritud piltidelt leitud väiksemad plekid
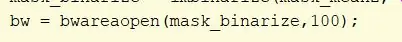
Eemaldage objektid, mille ühenduvus on väiksem kui 100 pikslit, kasutades funktsiooni mask_binarize funktsiooni bwareaopen ja määrake läviväärtuseks 100. Määrake muutujaks bw.
Samm: looge ketta struktureerimise element

Loo ketta struktureeriv element (raadiusega 2), kasutades funktsiooni strel. Määrake see se.
Samm 21: Tehke morfoloogilisi sulgemisoperatsioone

Võtke bw ja rakendage objektile morfoloogilise sulgemisoperatsiooni tegemiseks konstruktsioonielemendile liitmisfunktsioon.
Samm: leidke vähemalt 8 ühendatavad objektid
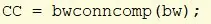
Võtke bw ja kasutage nuppu bwconncomp, et leida pildilt vähemalt 8 ühenduvusega objektid. Määrake numbriväljund väärtusele cc_1.
Samm: leidke ühendatud pikslite maksimaalne arv
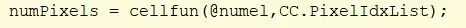
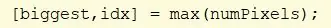
Kasutage funktsiooni cellfun funktsiooni CC numbrite täitmiseks igas CC lahtris. See leiab elementide arvu lahtris PixelIdxList. Määrake väärtus numbritele.
Leidke numPIxels maksimaalsed väärtused. Määrake suurim maksimum väärtusele „suurim” ja maksimaalse väärtuse indeks väärtusele „idx”.
24. samm: määrake maksimaalsed piksliväärtused väärtusele 0 ja otsige piksleid, mille ühenduvus on> = 26
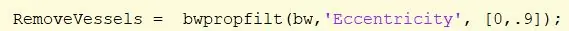
= 26 piksliühendust "src =" https://content.instructables.com/ORIG/FXY/DTW3/JEOIIEL4/FXYDTW3JEOIIEL4-p.webp

= 26 piksliühendust "src =" https://content.instructables.com/ORIG/FXO/GBX1/JEOIIELB/FXOGBX1JEOIIELB-p.webp

= 26 piksliühendus "src =" {{file.large_url | add: 'auto = webp & frame = 1 & height = 300' %} ">

= 26 piksliühendus "src =" {{file.large_url | add: 'auto = webp & frame = 1 & height = 300' %} ">
Seadke pildil “bw” suurimate väärtustega pikslid väärtuseks 0, muutes pikslid mustaks.
Leidke pildilt bwconncompi abil objektid, mille ühenduvus on vähemalt 26 pikslit. Määrake muutujale cc_1.
25. samm: eemaldage pildilt veresooned
Eemaldage pildil olevad veresooned, kasutades funktsiooni bwpropfilt vahemikuga [0, 0,9].
[0,9, 1] on välistatud, kuna 1 -le lähedased väärtused näitavad joont. Määrake jaotisele „Eemalda laevad”.
26. samm: joonise kuvamine
Kuvage iga filtreeritud pilt alamjoonisel. Imshow. piiride ja kitsaste sisenditega kuvab iga pildi alamjoonestruktuuris. Lisage igale pildile pealkiri, et eristada kasutatud filtrit.
Samm: eemaldage anumad ja loendage vereplekid
1. Võtke "RemoveVessels" ja rakendage piirkonnapropsis funktsiooni "Centroid", et tuvastada pildil olevate objektide keskpunktid. Need esemed peaksid vastama pildil esinevatele verehüüvetele.
2. Loendage tsentroidmaatriksi pikkuse järgi tuvastatud verehüüvete arv.
28. samm: diagnoosige võrkkesta pilt tuvastatud verehüüvete arvu alusel
Kasutage lausete abil pildi diagnoosimiseks tuvastatud verehüüvete arvu alusel.
Kui tuvastatud tsentroidide arv oli väiksem või võrdne 5, tuvastati pilt normaalseks.
Kui tsentroidide arv oli suurem kui 5, diagnoositi pildil diabeetiline retinopaatia.
Tulemus prinditakse käsuaknasse, kasutades fprintf.
Samm: kui on rohkem kui 5 plekki…
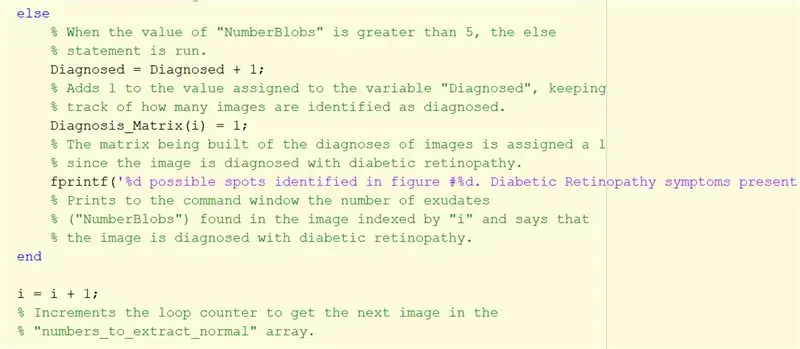
Korrake ülaltoodud juhiseid diagnoositud piltide jaoks muu avaldusena. See osa käivitub, kui plekke on rohkem kui 5.
Lõpetage if -lause.
Samm: korrake filtreerimisprotsessi normaalsete piltide jaoks, mille pildinumbri väärtused on 2 ja 3
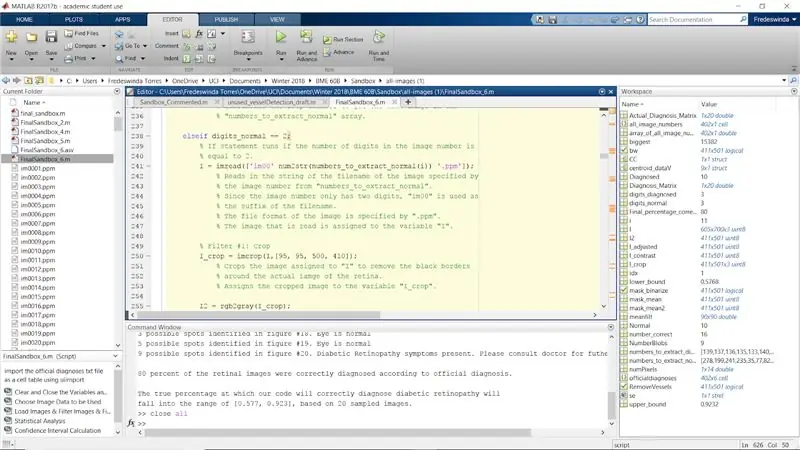
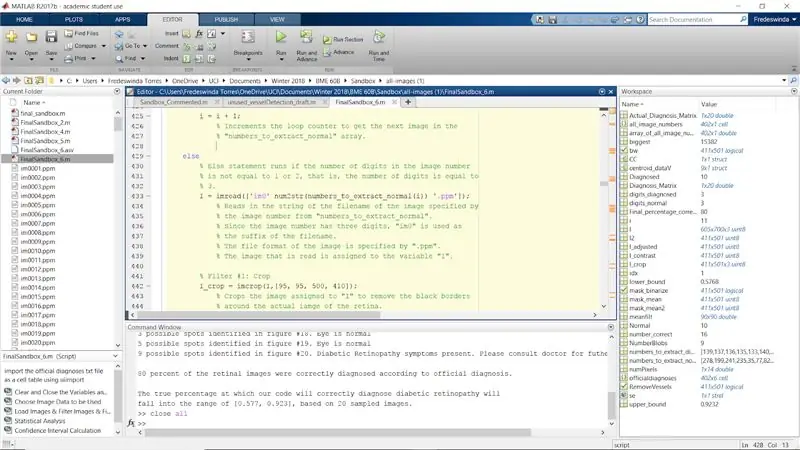
Korrake protsessi ülejäänud originaali puhul, kui laused, kui numel (numbrite arv pildi numbris) on võrdsed 2 ja 3. See lõpetab tavaliste piltide for -tsükli.
Lõpetage for -tsükkel.
Samm: korrake diagnoositud piltide jaoks kogu protsessi
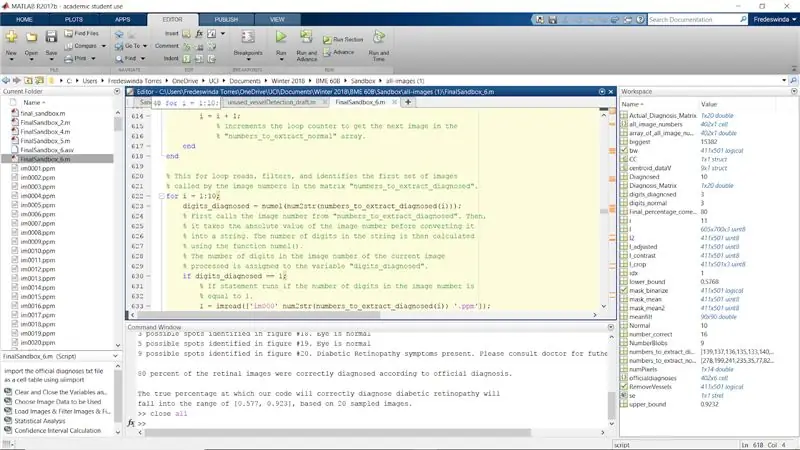
Korrake kogu protsessi, kasutades diagnoositud pilte, mis on loetletud maatriksis „numbrid_ekstrakti_diagnoositud”.
Vaadake kindlasti läbi kõik joonised (i) ja muutke need joonisteks (i+10), nii et diagnoositud arvud ilmuvad piltidena 11 kuni 20.
32. etapp: statistiline analüüs
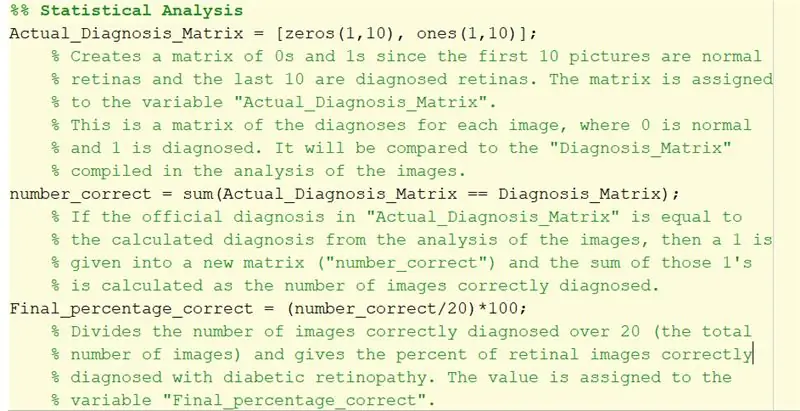
1. 'Tegelik_diagnoos_maatriks' kasutatakse tulemuste võrdlemiseks txt -failis leitud ametliku diagnoosiga. Esimesed 10 nulli näitavad, et esimesed 10 pilti peaksid olema normaalsed. Viimased 10 näitavad, et viimased 10 pilti tuleks klassifitseerida diabeetilise retinopaatia alla.
2. Kahekordne võrdusmärk, mida kasutatakse väärtuse „number_parandus” loomiseks, loob loogilise massiivi, kui võrrelda parameetri „Actual_Diagnosis_Matrix” vastavate elementide väärtust for -ahelast loodud väärtusega „Diagnosis_Matrix”.
Igale diagnoosile vastavale elemendile lisatakse 1, mis tähendab, et selle pildi õigesti diagnoositud kood. Kui see on vale, lisab see maatriksile 0.
Seejärel liidame selle summa kokku kõik. Teisisõnu, see leiab õigesti diagnoositud piltide summa.
3. „Lõplik_protsendi_õige” on arvutatud protsent sellest, kui täpne oli diagnoositud diabeetiline retinopaatia. Õigesti diagnoositud piltide arv jagatakse 20 -ga (piltide koguarv) ja korrutatakse 100 -ga, et leida edukate diagnooside protsent.
Samm 33. Usaldusintervalli leidmine
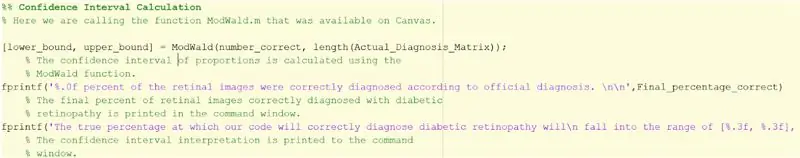
1. Veenduge, et olete alla laadinud ModWald.m, et seda funktsioonina nimetada. Ilma funktsioonita peaksite usaldatud intervalli ise arvutama, kasutades modifitseeritud Waldi meetodit.
2. Funktsioonil ModWald on 2 sisendit, kus esimene on õigesti tuvastatud piltide arv ja teine piltide koguhulk.
3. Funktsioon ModWald väljastab proportsioonide usaldusvahemiku alumise ja ülemise piiri proovivõetud andmete täpsuse huvides. Teisisõnu, annate protsentide intervalli, kus asub koodi täpsuse tõeline protsent.
4. Statistika ja usaldusintervalli käsuaknasse väljastamiseks kasutage allpool fprintf.
> fprintf ('%. 0f protsenti võrkkesta kujutistest diagnoositi ametliku diagnoosi kohaselt õigesti. / n / n', Final_percentage_correct)
> fprintf ('Tegelik protsent, mille korral meie kood diagnoosib diabeetilise retinopaatia õigesti, / n jääb vahemikku [%.3f, %.3f], tuginedes 20 valimipildile / n', alumine_seis, ülemine_seis)
Soovitan:
Juhtimine Blynk -rakenduse kaudu Nodemcu abil Interneti kaudu: 5 sammu
LED -i juhtimine Blynk -rakenduse kaudu Interneti kaudu Nodemcu abil: Tere kõigile, täna näitame teile, kuidas saate LED -i juhtida Interneti kaudu nutitelefoni abil
Klaasist kivist LED -toru (WiFi kaudu juhitav nutitelefonirakenduse kaudu): 6 sammu (koos piltidega)
Klaasist kivist LED-toru (WiFi kaudu juhitav nutitelefonirakenduse kaudu): Tere kaastegijad! Selles juhendatavas näitan teile, kuidas ehitada WiFi-juhitav LED-toru, mis on klaasikividega täidetud, et saada kena difusiooniefekt. Valgusdioodid on individuaalselt adresseeritavad ja seetõttu on mõned head efektid võimalikud
Äratus LAN -i kaudu igal arvutil traadita võrgu kaudu: 3 sammu

Äratus LAN -i kaudu igal arvutil traadita võrgu kaudu: Raspbpiani kujutise muutuste tõttu pole see õpetus enam ajakohane. Palun järgige värskendatud õpetust siit: https://www.instructables.com/id/Raspberry-Pi-As-Wake-on-LAN-ServerWOL on tänapäeval peaaegu kõigis Etherneti portides. See pole
Kuva tekst OLED -i kaudu Interneti kaudu: 9 sammu

Kuva tekst OLED -i kaudu Interneti kaudu: Tere ja tere tulemast! See lühike õpetus õpetab teid Magicblocks -i abil Magicbitil teksti kuvama. Selle eesmärgi saavutamiseks on kaks peamist meetodit; Kasutades süstimisplokki. Kasutades armatuurlauda. Kõigepealt logige sisse oma Magicb'i
Kõvakettad: diagnoosimine, tõrkeotsing ja hooldus: 3 sammu

Kõvakettad: diagnoosimine, tõrkeotsing ja hooldus: mis on kõvaketas?- Lihtsamalt öeldes salvestab kõvaketas kõik teie andmed. See sisaldab kõvaketast, kus kõik teie failid ja kaustad asuvad füüsiliselt. Teave salvestatakse kettale magnetiliselt, nii et see jääb kettaseadmesse ka siis, kui
